109906
论文已发表
注册即可获取德孚的最新动态
IF 收录期刊
- 3.4 Breast Cancer (Dove Med Press)
- 3.2 Clin Epidemiol
- 2.6 Cancer Manag Res
- 2.9 Infect Drug Resist
- 3.7 Clin Interv Aging
- 5.1 Drug Des Dev Ther
- 3.1 Int J Chronic Obstr
- 6.6 Int J Nanomed
- 2.6 Int J Women's Health
- 2.9 Neuropsych Dis Treat
- 2.8 OncoTargets Ther
- 2.0 Patient Prefer Adher
- 2.2 Ther Clin Risk Manag
- 2.5 J Pain Res
- 3.0 Diabet Metab Synd Ob
- 3.2 Psychol Res Behav Ma
- 3.4 Nat Sci Sleep
- 1.8 Pharmgenomics Pers Med
- 2.0 Risk Manag Healthc Policy
- 4.1 J Inflamm Res
- 2.0 Int J Gen Med
- 3.4 J Hepatocell Carcinoma
- 3.0 J Asthma Allergy
- 2.2 Clin Cosmet Investig Dermatol
- 2.4 J Multidiscip Healthc

基于 RT2 Profiler PCR 阵列和生物信息学分析对甲状腺乳头状癌 (PTC) 潜在生物标志物的探索
Authors Peng Y, Zhang HW, Cao WH, Mao Y, Cheng RC
Received 17 June 2020
Accepted for publication 24 August 2020
Published 28 September 2020 Volume 2020:12 Pages 9235—9246
DOI https://doi.org/10.2147/CMAR.S266473
Checked for plagiarism Yes
Review by Single anonymous peer review
Peer reviewer comments 2
Editor who approved publication: Dr Eileen O'Reilly
背景:近年来,甲状腺乳头状癌(PTC)发病率迅速增加,放疗、激素效应、基因突变等因素被认为与其发病密切相关。然而,PTC 的分子机制尚不清楚。因此,本研究拟结合 RT2 Profiler PCR 阵列和生物信息学方法,筛选出更准确的 PTC 关键基因和通路。
方法:首先通过 RT2 Profiler PCR 阵列分析 PTC 中差异表达基因(DEGs)。RT-qPCR 检测验证呈现显著差异表达的基因。使用 TCGA 数据库对扩展数据进行进一步验证。对差异基因进行了基因本体论(GO)和京都基因与基因组百科全书(KEGG)富集分析。为了构建蛋白-蛋白相互作用(PPI)网络,我们使用 STRING 和 Cytoscape 对这些 DEGs 进行模块分析。
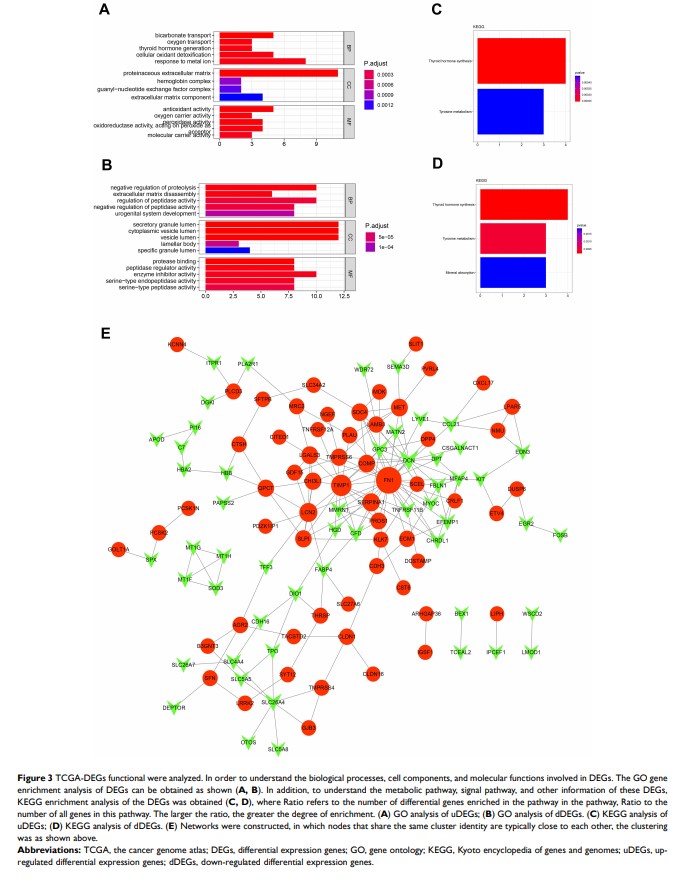
结果: RT2 Profiler PCR 序列显示 16 个差异表达基因,其中 13 个下调 DEGs(dDEGs), 3 个上调 DEGs(uDEGs), 13 个稳定的 DEGs 最终得到验证。TCGA 数据库中共有 155 个 DEGs,其中上调 DEGs(uDEGs) 82 个,下调 DEGs(dDEGs) 73 个。综合这两个结果,共提取 29 个重要基因,用 GO 和 KEGG 分析观察这些 DEGs 可能的作用机制。构建 PPI 网络,观察节点基因。预后分析进一步证实这些基因参与了 PTC 的生物学过程。
结论:本研究确定了一些潜在的分子靶点和信号通路,可能有助于提高我们对 PTC 发病机制的认识。
关键词:甲状腺乳头状癌;Qiagen RT2 Profiler PCR 阵列;生物信息学;DEGs;信号通路